Mit KI den Datendschungel an Spitälern durchforsten
ZHAW-Forschende stellen an der NeurIPS’24 in Vancouver, einer der wichtigsten KI-Konferenzen weltweit, ihre Arbeit vor. Sie befasst sich mit elektronischen Gesundheitsakten (electronic health records - EHRs) und deren Abfrage in natürlicher Sprache in verschiedenen Datenbanksystemen. Daraus ergeben sich neue Einsichten und Lösungen für die komplexe Aufgabe, natürliche Sprache in eine Abfragesprache für Datenbanken zu übersetzen.

Eine Herausforderung im Gesundheitssektor und anderen Bereichen ist, dass Informationen in mehreren verschiedenen Datenbankarchitekturen gespeichert werden. Daten können beispielsweise in Tabellen organisiert werden (relationale Datenbanksysteme), in Graphdatenbanken oder in Dokumentenspeicher. Um an spezifische Informationen zu kommen, werden Abfragesprachen wie SQL oder SPARQL genutzt. Die beiden Absolventen des Studiengangs Data Science an der ZHAW School of Engineering Sithursan Sivasubramaniam und Cedric Osei-Akoto erarbeiteten in ihrer Bachelorarbeit unter der Leitung von Jonathan Fürst eine neue multimodale Lösung, um verschiedene Systeme und Abfragesprachen in Bezug auf ihre KI-Tauglichkeit zu vergleichen. Die Arbeit beantwortet zum ersten Mal die Frage, wie gut Large Language Models (LLMs) wie ChatGPT, Llama3 oder Gemini von natürlicher Sprache in die jeweilige Datenbanksprache übersetzen können. Aus der Bachelorarbeit wurde unter Mitarbeit der ZHAW-Forscher Yi Zhang und Kurt Stockinger eine Publikation an der renommierten NeurIPS Konferenz in Vancouver.
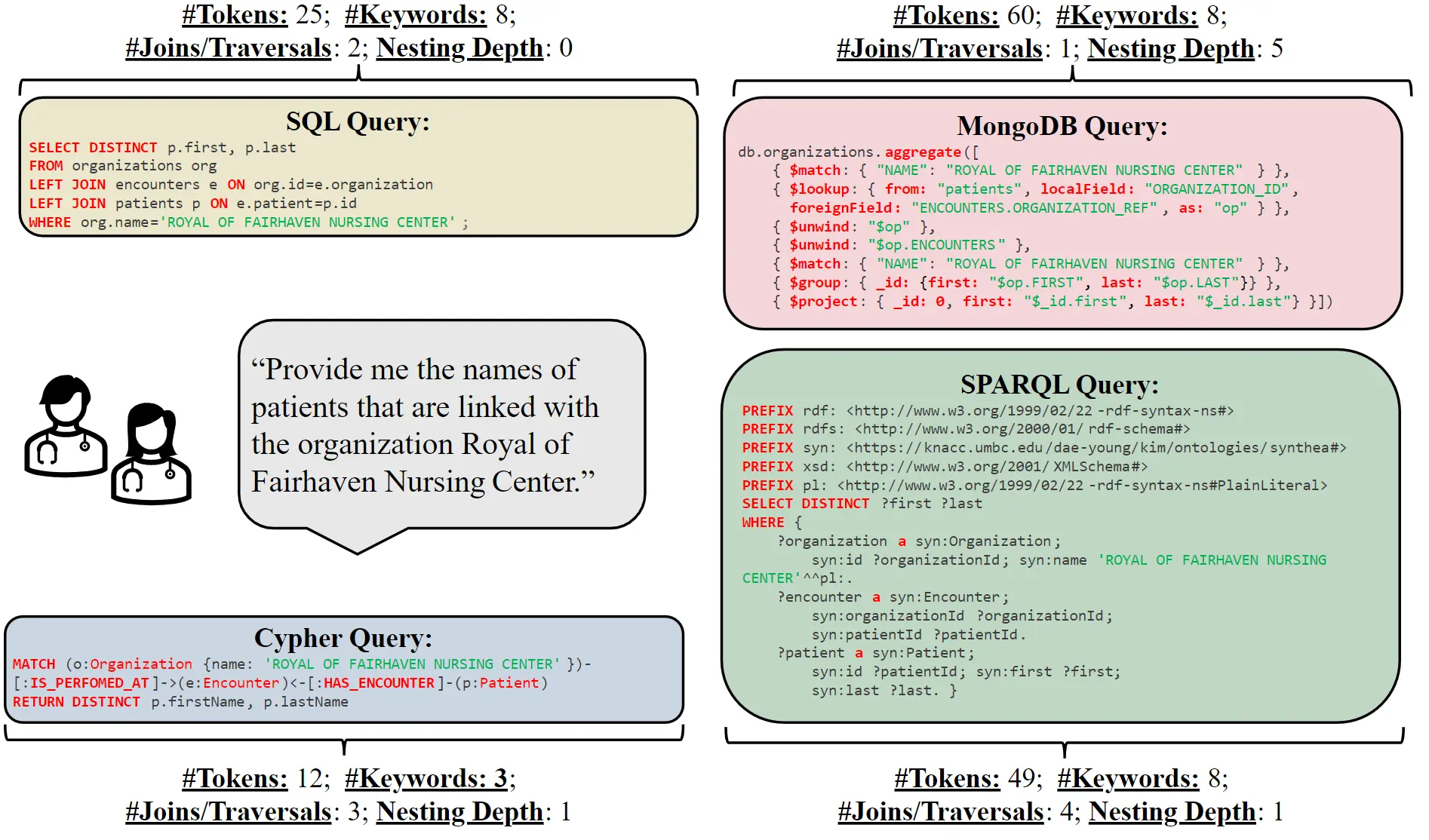
Erstmals ein Benchmark für unterschiedliche Datenbankarchitekturen
Das Herzstück der Forschung ist der SM3-Text-to-Query Benchmark, der als erster seiner Art mehrere Datenbankarchitekturen und Abfragesprachen integriert. Der Titel des Benchmarks setzt sich aus den 3 «Ms» zusammen (multi-modal und medizinisch). Text-to-Query bedeutet, dass ein Text in natürlicher Sprache eine Abfrage in der Datenbank startet, z.B. «Finde die Patientennamen mit Symptomen ABC im Spital XYZ». Basierend auf synthetischen, also automatisch generierten und daher datenschutzrechtlich unbedenklichen, Patientendaten und einer umfassenden Gesundheitsterminologie (SNOMED-CT Taxonomie), unterstützt dieser Benchmark verschiedene Datenbankarchitekturen. Dies ermöglicht eine umfassende Evaluation der Leistungsfähigkeit verschiedener Systeme.

Innovative Methodik und umfassende Evaluation
Die Forschenden entwickelten 408 Fragenvorlagen, die zu einem umfangreichen Benchmark-Set von 10’000 natürlichen Sprach-/Abfragepaaren für jede Abfragesprache erweitert wurden. Diese Abfragesprachen umfassen vier bekannte Sprachen (SQL, MQL, Cypher und SPARQL). Das Ziel der Experimente mit diesen Daten war, herauszufinden wie gut grosse Sprachmodelle (LLMs) natürliche Sprache in diese vier verschiedenen Abfragesprachen übersetzt. Zwei offene (beide LLama von Meta) und zwei geschlossene Sprachmodelle (Gemini von Google und ChatGPT von OpenAI) wurden analysiert.
Ergebnisse und Ausblick
Die Ergebnisse der Studie zeigen deutliche Unterschiede in der Leistung der verschiedenen Datenbankmodelle und Abfragesprachen. In Zukunft soll der Benchmark auf zusätzliche Abfragesprachen oder reale, standardbasierte Patientendatenbanken angepasst werden. Dies macht den Benchmark zu einem wertvollen Werkzeug für die zukünftige Forschung und Entwicklung im Bereich der digitalen Gesundheitsversorgung.
Mit ihrer Arbeit setzen die Forschenden neue Massstäbe in der Bewertung von Text-zu-Abfrage-Systemen. Dieser standardisierte und datenschutzfreundliche Benchmark bietet nicht nur der wissenschaftlichen Gemeinschaft, sondern auch der breiten Öffentlichkeit wertvolle Einblicke und Lösungen für die Herausforderungen der modernen Gesundheitsversorgung.
Weitere Informationen
- Konferenz NeurIPS: 2024 Conference
- Paper: https://doi.org/10.48550/arXiv.2411.05521
- Code and data: https://github.com/jf87/SM3-Text-to-Query