Projektbeispiel: Bayessche Analyse von NMR-Spektren
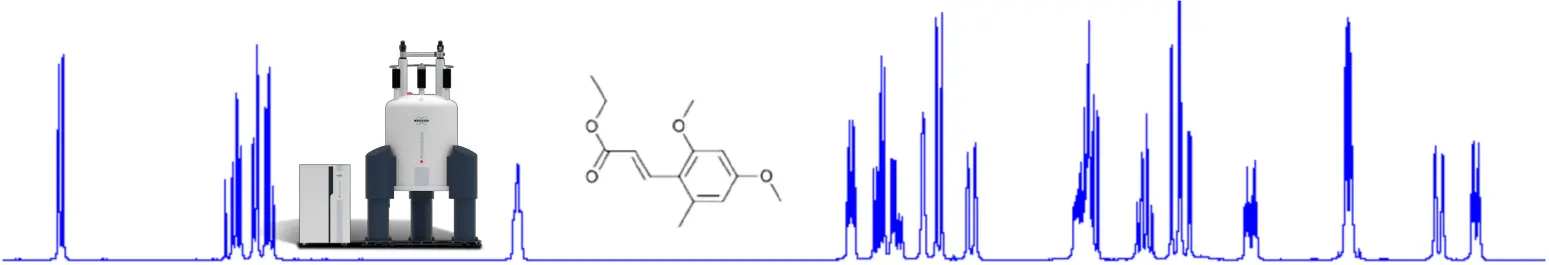
Im Rahmen des Innosuisseprojekts Maschinelles Lernen für NMR-Spektroskopie hat der Schwerpunkt WRA in Zusammenarbeit mit Bruker BioSpin eine Sequentielle Monte-Carlo Sampling (SMC) Methode zur Analyse von NMR-Spektren entwickelt.
Das Ziel dabei ist für ein gemessenes NMR-Spektrum und gegebene Molekülstruktur (genauer die Struktur des entsprechenden Spinsystems) die Strukturparameter (sog. chemische Verschiebungen und Kopplungskonstanten) zu bestimmen.
Unser in C++ entwickeltes Tool kombiniert eine quantenmechanische Simulation des NMR-Spektrums (für eine Wahl von Spinsystem-Parametern) mit einem SMC Sampler (mit HMC-Kernel und erweitertem Likelihood-Tempering), Vorwissen über das Spinsystem wird dabei mittels Prior-Verteilung modelliert. Unter Einbezug des gemessenen NMR-Spektrums wird schrittweise die Posterior-Verteilung konstruiert und die Spinsystem-Parameter bestimmt. Eine ausführlichere Beschreibung ist unter https://authors.elsevier.com/sd/article/S1090-7807(24)00107-1 zu finden.
Die folgende Animation illustriert den Sampling-Prozess anhand eines einfachen Testsystems (AA'-BB'-Spinsystem) mit bekannten Zielgrössen (dargestellt als grüne vertikale Linien). Die Anfangsverteilung (Prior-Verteilung) ist breit, die chemische Verschiebungen (obere 2 Plots) und Kopplungskonstanten (untere Plots) sind kaum bestimmt. Unter Einbezug der NMR-Daten konzetrieren sich die Verteilungen mit fortschreitendem Sampling-Prozess zunehmend um die Zielgrössen. Die Posterior-Verteilung nach Abschluss des SMC-Temperings ist schliesslich ein schmale Verteilung um die jeweiligen Zielgrössen.
